湾豆说:近日,陈鹏/樊新元团队在J. Am. Chem. Soc.上发表研究论文,将IEDDA生物正交剪切反应进一步应用到蛋白质-DNA作用界面的化学调控,利用四嗪触发反式环辛烯的脱除实现Cas9蛋白质与PAM序列结合的调控,再通过融合脱氨酶APOBEC1发展出了活细胞中化学可控的C到T碱基编辑技术BaseBAC (Bioorthogonally ACtivatable Base Editor),用于原位调控GSDME蛋白质的自抑制性C端表达 (Q287位CAA到TAA的碱基编辑),用于细胞焦亡的化学调控。
生物正交反应是指可以在生物体系中进行、且不会与天然生物化学过程相互干扰的一类化学反应。初期的生物正交反应主要是指偶联反应,主要用于在生物体复杂环境中对目标分子进行标记、示踪、修饰改造等。随着研究的深入和应用的拓展,生物正交反应的内涵也在不断拓展,在最初的偶联反应基础上,拓展了剪切反应等新类型。北京大学化学与分子工程学院陈鹏/樊新元团队一直致力于生物正交剪切反应的开发与应用研究 [1],并结合蛋白质的基因密码子扩展技术,实现了基于四嗪/反式环辛烯、反式环辛烯/叠氮苄基、铜/炔丙基等一系列生物正交剪切反应对的发展优化及蛋白质原位激活与功能调控的技术开发。其中,四嗪/反式环辛烯的逆电子需求的Diels–Alder反应(IEDDA)因其高特异性、高反应速率等优点而得到广泛应用。
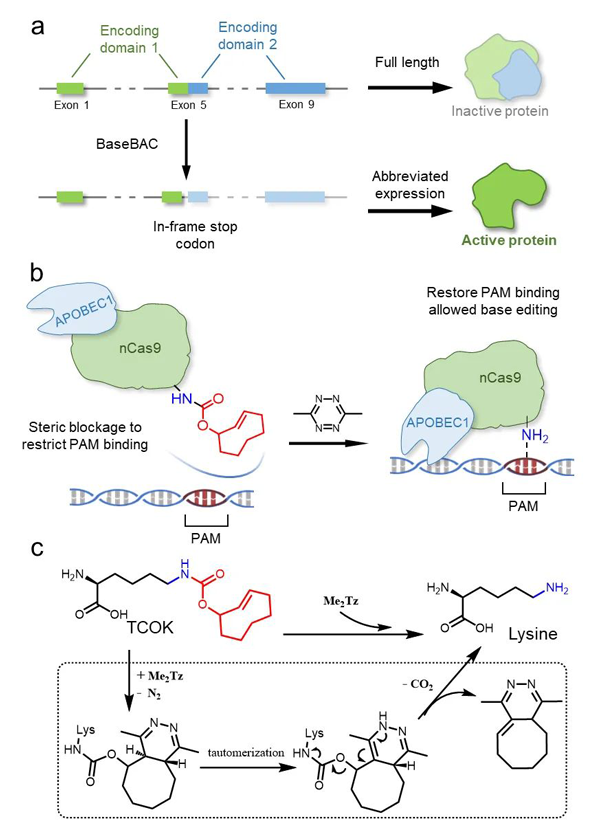
图1. 基于生物正交脱笼的BaseBAC碱基编辑技术示意图
细胞焦亡是由gasdermin(GSDM)家族蛋白在经过蛋白酶剪切掉C端结构域后,其N端结构域释放并在细胞膜上寡聚打孔而产生的一种细胞死亡方式 [2,3]。因此,通过控制C端结构域的表达可以实现GSDM蛋白的活性调控。近年来,研究人员通过在半催化失活的nCas9上融合胞嘧啶脱氨酶(APOBEC1),开发出了可以对目标碱基进行从C到T转化的胞嘧啶碱基编辑器(CBE)[4],可以用于在开放阅读框中引入终止密码子从而实现目标蛋白质C端结构域的表达调控。
陈鹏/樊新元团队据此开发了基于生物正交剪切反应的CBE激活技术BaseBAC。CBE的靶向依赖于其nCas9结构域,而Cas9需要先识别PAM序列后才会进行底物序列的查验。因此,作者首先对Cas9的晶体结构进行分析,K1200位点处于与PAM序列相互作用的界面。如将K1200替换为非天然氨基酸TCOK,TCO基团的疏水性与空间位阻将可能阻止Cas9与PAM序列的结合。只有在加入小分子激活剂Me2Tz后,发生IEDDA反应使TCO基团脱除,产生出原来的赖氨酸并恢复蛋白的活性。作者在全催化失活的dCas9中的K1200位点插入TCOK,在加入Me2Tz后可观察到目的基因的敲低表达,验证了通过干扰Cas9与PAM序列结合来调控基因表达的设想。然后,作者在CBE中nCas9结构域的K1200位点插入TCOK,生成可原位激活碱基编辑的BaseBAC,并将此靶向基因组中的EMX1基因,展示了BaseBAC时间特异地对基因进行精准的碱基编辑。
作为应用展示,作者设计了靶向GSDME的C端与N端之间序列的guide RNA,导向BaseBAC编辑GSDME 287位的谷氨酰胺。当加入Me2Tz激活BaseBAC后,该密码子从CAA修改成TAA,GSDME提前中止翻译而只表达活性N端,从而触发细胞焦亡,进而引起肿瘤免疫等一系列下游反应。最后,作者在活体小鼠体内进行BaseBAC对GSDME的碱基编辑,展示了TCOK—Me2Tz脱笼反应的生物兼容性,以及在活体动物上进行时间特异碱基编辑的可行性。
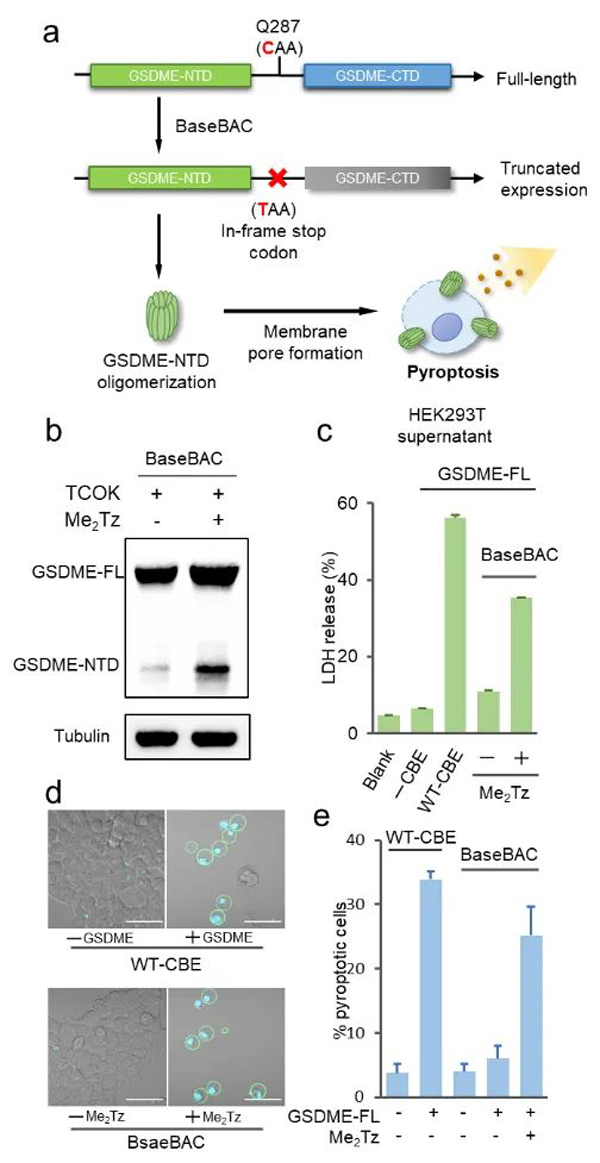
图2. 使用BaseBAC对GSDME进行功能性截短并原位产生细胞焦亡
综上所述,该研究开发出生物正交激活的碱基编辑技术BaseBAC,在细胞以及活体动物上展示了原位激活碱基编辑所触发的细胞焦亡。此激活技术针对Cas9对底物DNA的结合而开发,理论上适用于各种Cas9衍生的基因编辑工具中,包括Adenosine Base Editor以及Prime Editor等。另一方面,细胞中含有多种在表达时为自抑制状态的基因,其功能亦可以通过编程BaseBAC而进行时间特异地激活。Me2Tz介导的IEDDA反应具有良好的生物兼容性,使其能应用于多种生物学研究场景,具有广阔的应用前景。
北京大学化学与分子工程学院、北大-清华生命科学联合中心、深圳湾实验室资深研究员陈鹏教授和北京大学化学与分子工程学院樊新元副研究员为该论文的共同通讯作者,深圳湾实验室博士后魏书程、北京大学杨绍君以及曾祥梅为该论文的共同第一作者。该工作得到了国家自然科学基金委、科技部、北京分子科学国家研究中心以及北大-清华生命科学联合中心的资助。
参考文献:
[1] Wang, J.; Wang, X.; Fan, X.; Chen, P. R., Unleashing the Power of Bond Cleavage Chemistry in Living Systems. ACS Cent. Sci. 2021, 7 (6), 929-943.
[2] Shi, J.; Zhao, Y.; Wang, K.; Shi, X.; Wang, Y.; Huang, H.; Zhuang, Y.; Cai, T.; Wang, F.; Shao, F. Cleavage of GSDMD by Inflammatory Caspases Determines Pyroptotic Cell Death. Nature 2015, 526 (7575), 660.
[3] Kesavardhana, S.; Malireddi, R.; & Kanneganti, T. D. Caspases in Cell Death, Inflammation, and Pyroptosis. Ann. Rev. Immunol. 2020, 26 (38), 567–595.
[4] Komor, A. C.; Kim, Y. B.; Packer, M. S.; Zuris, J. A.; Liu, D. R. Programmable Editing of a Target Base in Genomic DNA without Double-stranded DNA Cleavage. Nature 2016, 533 (7603), 420.
论文标题:
Bioorthogonally Activatable Base Editing for On-Demand Pyroptosis
文章来源 | X-MOL

